Proteinmodifikationen – Acetylierung, Ubiquitinylierung, Phosphorylierung, SUMOylierung
Proteinmodifikationen – Acetylierung, Ubiquitinylierung, Phosphorylierung, SUMOylierung
Definition: Kovalente Modifikation
Bei einer kovalenten Bindung teilen sich die reagierenden Atome ein Eltronenpaar ("Valenzelektronen") und gehen somit eine chemische Bindung ein. Diese Prozess wird meist durch Enzyme katalysiert. Das Ergebnis ist eine kovalente Bindung bzw. Modifikation
Das kovalente Anhängen eines anderen Moleküls kann die Aktivität eines Enzyms oder anderen Proteinen modifizieren. In diesen Fällen stellt ein Spender Molekül einen funktionellen Teil, der die Eigenschaften des Enzyms ändert. Die meisten Modifikationen sind reversibel. Man spricht auch von einer reversiblen kovalente Modifikation.. Phosphorylierung und Dephosphorylierung sind die geläufigsten kovalenten Modifikationen in biologischen Systemen. Daneben gibt es aber noch weitere, z.B. Acetylierung, Ubiquitinylierung und SUMOylierung.
Arten von Modifikationen
Acetylierung
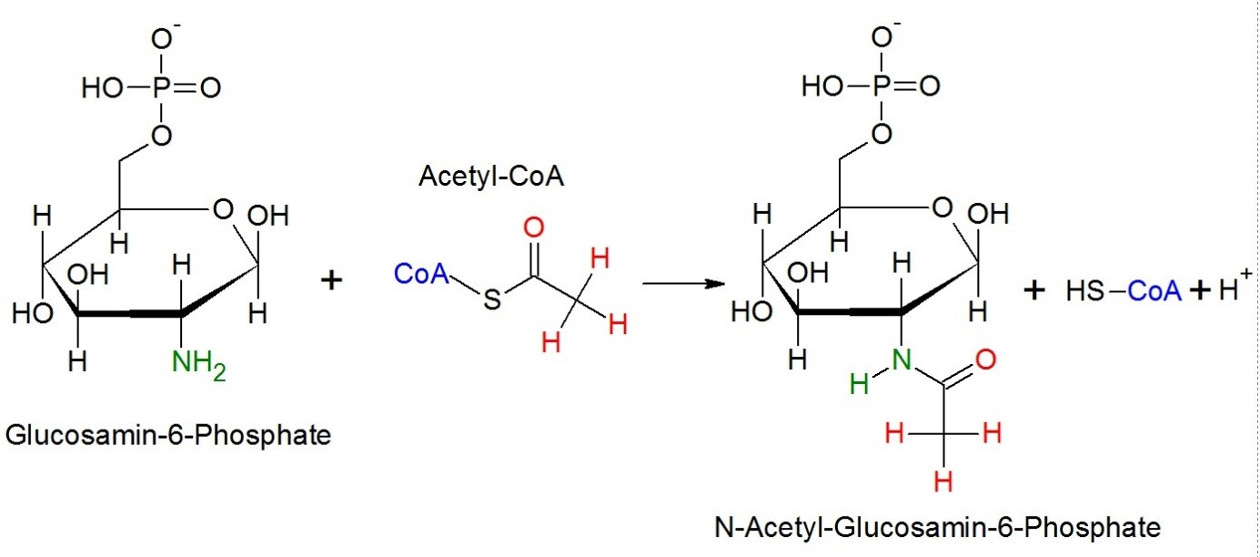
Abbildung 1: Acetylierung am Beispiel von Glucosamin-6-Phosphate. CoA fungiert als Acetyltransferase und überträgt seine Acetylgruppe auf den Aminorest des Glucosamins.
Acetylierung und Deacetylierung spielen eine wichtige Rolle bei der Transkription von Genen. Konkreter gesagt, acetylierte Histone stehen in Verbindung mit aktiv transkribierten Genen, wohingegen methylierte Genabschnitte nicht transkribiert werden. Acetylierte und methylierte Genabschnitte dienen als Erkennungssequenzen für RNA- und DNA-Polymerasen.
Die Acetylierung und Desacetylierung von Proteinen reguliert somit deren Funktion.
Dabei handelt es sich um eine posttranslationale Proteinmodifikation. Eine Acetylase übertägt eine Acetylgruppe von Acetyl-Coenzym A auf das Zielprotein, meist auf ein Lysin.
Die Deacytlase kann die Acetylgruppe wieder entfernen und macht die Protein-Acetylierung somit reversibel.
Phosphorylierung
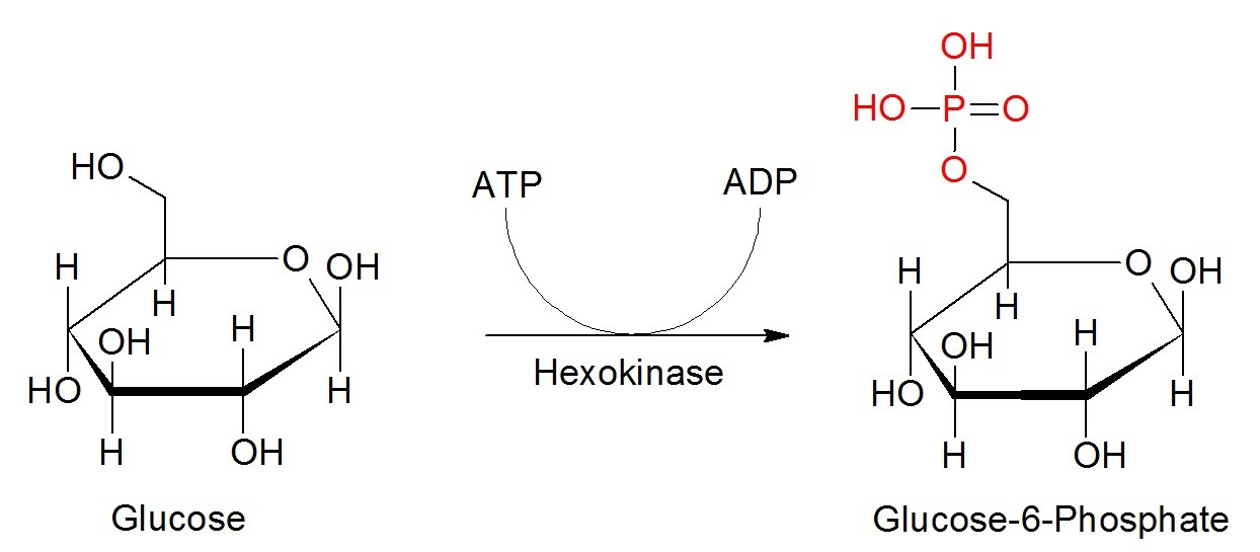
Abbildung 2: Phosphorylierung am Beispiel von Glucose. Glucose wird mit Hilfe der Hydrolyse von ATP zu ADP am C6-Atom durch eine Hexokinase phosphoryliert.
Phosphorylierungen dienen in der Regel zur Aktivierung und Regulation von Molekülen, meistens Enzymen.
Hierbei wird eine Phosphatgruppe durch eine Phosphatkinase an ein organisches Molekül angehangen.
Im Falle von Proteinen, entstehen somit Phosphoproteine, die in unserem Nervensystem eine zentralle Rolle spielen. Bekannte Beispiele, die durch Phosphorylierung reguliert werden sind Enzyme (z.B. Rezeptor-Tyrosin-Kinasen, MAP-Kinasen) und Transkriptionsfaktoren (z.B. c-Jun, CREB).
Ubiquitinylierung
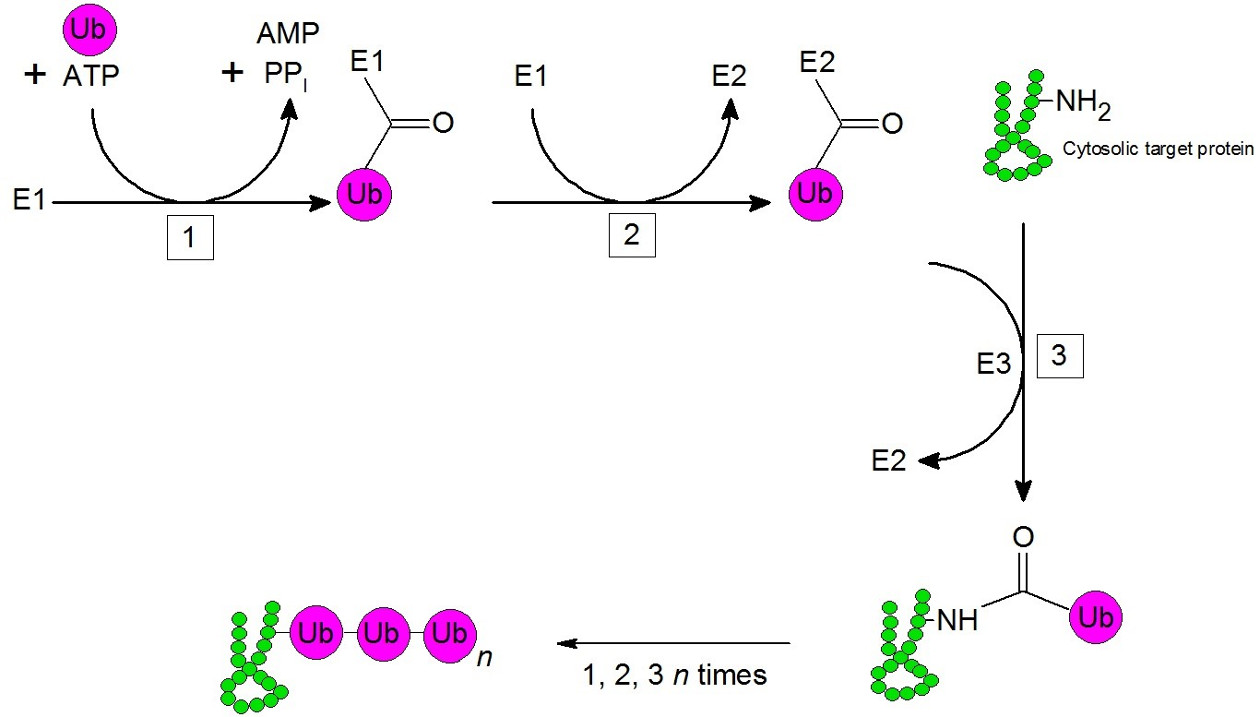
Abbildung 3: Ubiquitinylierung am Beispiel eines zytosolischen Proteins. 1.) Enzym E1 aktiviert in einem ATP-abhängigen Schritt Ubiquitin. Danach wird Ubiquitin auf E2 übertragen. Eine Ubiquitin Ligase (E3) überträgt das gebundene Ubiquitin auf die Aminogruppe (-NH2) eines Lysins des abzubauenden Proteins. Zusätzliche Ubiquitinmoleküle werden dem abzubauenden Protein durch wiederholen der Schritte 1-3 hinzugefügt. Das polyubiquitinylierte Protein erhält so die Erkennungssequenz für das Proteasom und die damit verbundene Degradation.
Wird Ubiquitin auf ein Zielprotein übertargen spricht man von der Ubiquitinierung . Sie hat zur Folge, dass das Protein im Proteasom abgebaut wird.
Außerdem wird zwischen der Mono-(einzelnes Ubiquitin-Molekül) und Polyubiquitinierung (Ubiquitinketten) von Proteinen differenziert.
SUMOylierung
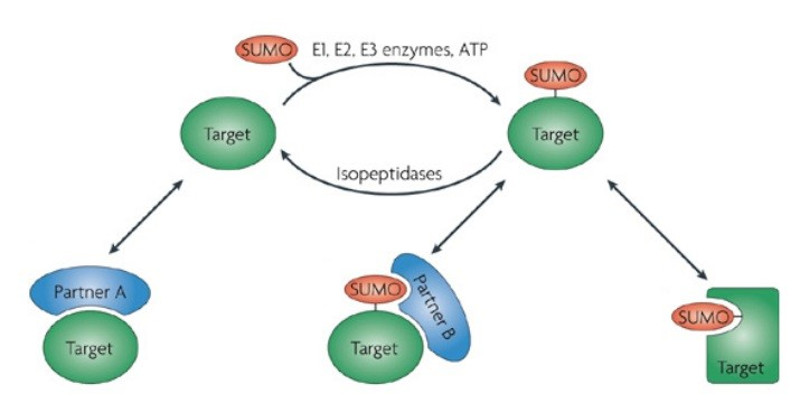
Abbildung 4: SUMOylierung
Das kovalente anhängen von SUMO-Proteinen (Small Ubiquitin-related MOdifier) an die Lysinreste von Proteinen wird als SUMOylierung bezeichnet. Die Umkehrung dieser kovalente Modifikation heißt "DeSUMOylierung".
Angebote von Hölzel Diagnostika
Signal Seeker™ Kits und Signalweg-Werkzeuge:
Signal Seeker™ Kits verfolgen die Absicht Forschern die Entdeckung neuer Signalwege, welche dynamische Protein Modifikationen, wie Ubiquitinylierung, SUMOylierung und Phosphorylierung beinhalten, zu ermöglichen. Die Stärke der Signal Seeker™ Kits liegt in der Optimierung von Reagenzien, welche die Detektion von Proteinmodifikationen im Nanogramm-Bereich ermöglichen. Das BLASTR™ Buffer System ermöglicht es dem Anwender außerdem aus einem einzigen Extrakt verschiedene Proteinmodifikationsänderungen zu untersuchen. Signal-Seeker™ Kits bieten zudem eine anwenderfreundliche Einführung zu der oft technisch komplexen Welt von dynamischen Signalweg Regulationsmechanismen. Die detaillierten Kit Manuals sind eine große Informationsquelle für Anwender. Einige Kit Reagenzien, die Affinitäts-Beads und Antikörper enthalten, sind auch separat erhältlich. Insbesondere die Ubiquitin Affinity Beads sind wesentlich besser als andere herkömmlich zu erwerbende Affinitäts-Beads, da sie eine Affinität für alle ubiquitinylierten Proteine, einschließlich mono-ubiquitinylierter Natur, vorweisen. In der Literatur werden zwei Referenzen einer frühen Produktentwicklung zitiert.
Ausgewählte Produkte
- Signal Seeker™ Phosphotyrosine Enrichment Kit 30
- Signal Seeker™ Ubiquitin Enrichment Kit 30
- Signal Seeker™ Sumoylation Enrichment Kit 30
Literatur
- Stryer et al. Biochemistry 5th Edition, Ch. 10.4, p. 423
- Anti-phosphorylation affintiy beads (Cat. # BK160), Law A. et al. 2015. Temporal regulation of phosphotyrosine-modified Rac1 in response to epidermal growth factor (EGF) stimulation. American Society for Cell Biology, 2015, Poster P2126.
- Anti-acetyl-lysine monoclonal antibody (Cat. # AAC01), LaBarge et al. 2015. p300 is not required for metabolic adaptation to endurance exercise training. The FASEB Journal article doi: 10.1096/fj.15-281741. This paper uses anti-acetyl lysine to probe extracts of transgenic mouse muscle tissue, looking for the effects of a knocked out HDAC gene.



